
QbSyst2e.9.html
Système du Québécium
Suivant QbSyst2e.10.html
Précédent
QbSyst2e.8.htmlDébut QbSyst2e.1.html
Chapitre 5
Applications aux biomolécules
Les dérogations
au code biomoléculaire normal

Fig. 89. Le code biomoléculaire normal. (Code cb1 de la figure 61).
Normalité et dérogations.
Dans le présent chapitre, nous poursuivons l'étude des variantes du code biomoléculaire en les considérant comme des dérogations à un code que nous appelons normal et nous y ajoutons les autres dérogations qui nous sont connues.
Nous convenons de prendre comme code biomoléculaire normal celui qui apparaît à la figure 89.
Nous allons voir que certains codes cb1 à cb23 doivent subir des corrections. Les codes corrigés correspondants sont cb1+ à cb23+.
Nous allons essayer de comprendre dans quelle mesure ces dérogations s'adaptent au système du québécium. On peut reconnaître 4 classes de dérogations. Tableau 37.
Tableau 37. Les classes de dérogations.
1re classe : préserve les 64 codons et les 20 acides aminés décrits. Comprend essentiellement les 16 codes cb2 à cb23.
2e classe : ajoute des acides aminés nouveaux.
3e classe : ajoute en plus des codons nouveaux.
4e classe : couvrirait les synthèses de protéines qui se feraient sans l'intervention de codons et en dehors de la traduction qu'ils déterminent.
1re classe de dérogations.
Dans cette classe, les codons ont une affectation autre que dans le code normal. Les données sont rassemblées dans une figure basée sur la strate 4 annotée. Les affectations des codons sont énumérées en regard de ceux-ci; nous ne figurons pas les strates 1 et 2, où les 20 acides aminés restent inchangés; nous n'avons pas inscrit les redondances et les multiplicités. Figures 90, 91, 92, 93.

Fig. 90. Dérogations de 1re classe. Strate 4 du code universel sur laquelle on a mis en évidence les cases malléables et certaines cases stables. On indique les 43 dérogations au code universel avec le code où chacune se manifeste.
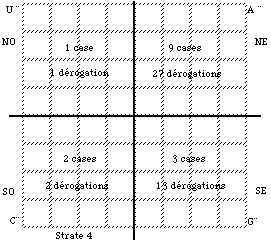
Fig. 91. Dérogations de 1re classe. Répartition dans les quadrants de la strate 4.
Dans ces strates, 15 cases acquièrent au moins une fois une affectation autre que dans le code normal, nous les appelons malléables. Elles sont mises en évidence dans la figure 112. Nous relevons 43 cas de changement d'affectation d'une case. Deux particularités s'imposent à l'attention.
...................
Les codons GXX ayant G comme 1re lettre sont tous stables....................
Les codons ayant A comme 3e lettre sont particulièrement malléables.Ainsi, les cases malléables se pressent dans le quadrant NE. Figure 92.
Voici la répartition des dérogations par case. Tableau 38.
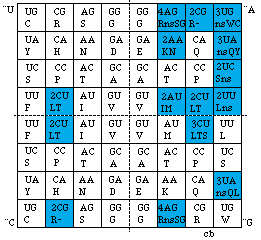
Fig. 92. Dérogations de 1re classe. Dégénérescences dans les codes biomoléculaires cb1 à cb23, strate 4. On a marqué les affectations.
Tableau 38. Dérogations de 1re classe dans les 16 codes cb1 à cb23 (et de 2e classe). Cases de la strate 4.
1re colonne : affectation normale et redondance normale
CUUL6
...3TCUCL6
...3TCGCR6
...3-AGCS6
...21SAGAR6
...2ns...5S...9S...13G...14G...21SCGAR6
....3-UGAns3
....
2W...3W...4W...5W...9W...10C...13W...14W...21W...(1SeC)AAAK2
...9N...14NUAAns3
...6Q...14YUCAS6
...22nsAUAI3
...2M...3M...5M...13M...21MCUAL6
...3TUUAL6
...23nsAUGM1
...(2fM)CUGL6
...3T...12SUAGns3
...6Q...15Q...16L...22L...(1pK)AGGR6
...2ns...5S...9S...13G...14S...21S15 cases sont affectées (+1). 43 dérogations sont constatées (+3).
--------------------------
1re classe :
N. de dérogations
..N. de cases...............Total1re classe
1
..............................7.............................72
..............................3.............................63
...............................0.............................04
..............................1.............................45
..............................1..............................56
..............................2.............................129
..............................1..............................9Total
......................16.............................43--------------------------
2e classe :
N. de dérogations
..N. de cases...............Total1
..............................3.............................(3)Les codons dégénérés.
Les cases de la strate 4 munies de dérogations sont en dégénérescence par rapport aux affectations, par exemple, elles correspondent à plus d'un acide aminé. Ainsi, la case AUC n'a qu'une affectation, désignée par la lettre I, elle a la dégénérescence 1. La case CUG a 3 affectations : L, T et S, elle a la dégénérescence 3. Ainsi, 15 cases de la strate du code biomoléculaire ont une dégénérscence supérieure à l'unité. Figure 92, Tableau 39.
Tableau 39. Dégénérescences des codons de la strate 4 des codes biomoléculaires cb1 à cb23
Il y a 22 affectations possibles, dont 20 acides aminés normaux, ns et le silence.
Nombre d'affectations
..............Dégénérescences
.........Codons1
.............................492
..............................93
..............................44
..............................2Total
........................642e classe de dérogations.
Dans cette classe, des codons ont une affectation autre que dans le code normal et codent pour des acides aminés nouveaux.
Placement dans les strates 1 et 2. Les strates 1 et 2 ont 20 cases. Si nous avons plus de 20 acides aminés à loger, il faut user de conventions : ou bien nous logeons les acides aminés excédentaires n dehors de la grille de 20 cases, ou bien nous logeons plus d'un acide aminé dans certaines cases desdites cases.
Nous optons pour la 2e hypothèse : certaines cases des strates 1 et 2 contiendront plus d'un acide aminé. Nous essayerons de mettre ensemble dans une même case des molécules paraissant compatibles. C'est là pour le moment une pure convention, facilitant la présentation des résultats. Il faudra examiner par la suite si les molécules ainsi associées auraient une entité unique en commun.
Acides aminés naturels. Nous examinons le cas de 4 acides aminés naturels.

Fig. 93. Cystéine et cystine.
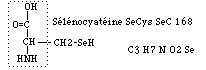
Fig. 94. Sélénocystéine.

Fig. 95. Codage de la sélénocystéine par le codon UAG.
Cystine. La cystine est mentionnée ici pour mémoire. Selon le souvenir de l'auteur, on l'a déjà placée dans la liste des codés. En fait, on la trouve dans les protéines mais elle n'est pas codée directement par un codon. Elle provient de la cystéine.
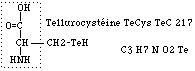
Fig. 96. Tellurocystéine. (Hypothétique).
La cystéine est un acide aminé thiol codé par les codons UGU et UGC dans tous les codes et par UGA dans le code cb10. 2 molécules de cystéine d'une protéine se condensent en cystine par un doublage à la suite d'une oxydation avec formation d'une molécule d'eau et établissement d'une liaison entre 2 atomes de soufre. Figure 93.
Cette transformation intervient effectivement pour établir des ponts disulfure SS et des boucles dans les structures secondaires des protéines.
2 C3H7NO2S + O = H2O + C6H12N2O4S2
Sélénocystéine. La sélénocystéine SeC est un acide aminé sélénol ou thiol substitué, avec substitution du sélénium à la place du soufre. Figure 94.
Elle apparaît en réponse à un codon UGA le plus souvent traduit pas un stop, dans le code cb1 devenant cb1+ touchant les protéines humaines, celles des animaux, des Archaea et des Eubactéries. Le code cb1+ renferme donc 21 acides aminés.
Sa formation dépend du codon UGA et de plusieurs autres placés en aval dans l'ARNm. Elle requiert la formation d'éléments en boucles dénommés SECIS. Figure 95.
Elle semble jouer un rôle dans le cancer, le sida et la maladie d'Alzheimer.
Co-traduction et post-traduction. L'apparition de SeC se fait par la traduction habituelle et passe par l'intervention d'un anticodon spécifique. Certains l'ont cependant qualifiée de processus co-traductionnel. Les processus post-traductionnels sont nombreux et donnent par exemple l'hémoglobine.
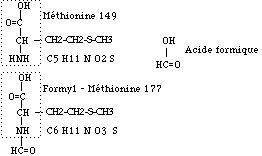
Fig. 97. Formyl-méthionine.
Il est naturel de placer la sélénocytéine SeC dans la même case que la cystéine C.
Tellurocystéine. On a synthétisé des acides aminés et des protéines contenant du tellure. Par extrapolation, on peut prédire la découverte dans les organismes de mécanismes capables d'en fabriquer et d'en insérer dans les protéines, mettant en jeu, par exemple, le codon UGA et la tellurocystéine TeC, acide aminé tellurol homologue de la sélénocystéine SeC et de la cystéine C. Figure 96.
La formyl-méthionine. La formyl-méthionine fM résulte de l'addition d'un groupement CO à la souche de la molécule de méthionine, sans changement de son radical. Figures 52, 97.
Elle figure dans le code cb2+ en réponse au codon AUG et elle sert d'initiateur à la place de la méthionine du code normal. Aussitôt apparue au début d'une chaîne protéique, elle perd son radical formyl et devient de la méthionine normale et celle-ci disparaît à son tour.
Le code cb2+ peut faire apparaître de la méthionine à l'intérieur d'une protéine, mais c'est un codon différent qui en commande la présence : AUA.
La formyl-méthionine et non la méthionine sert d'initiateur chez les procaryotes.
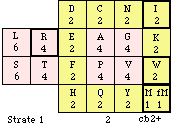
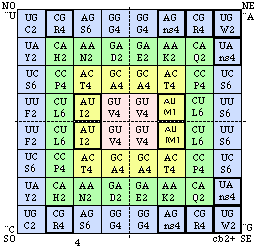
Fig. 98. Code biomoléculaire cb2+ des mitochondries des Vertébrés. Comme le code universel sauf altération de 18 cases mises en évidence. 60 cases codantes pour les acides aminés.
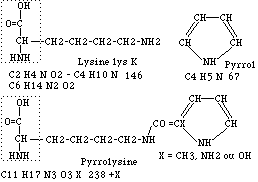
Fig. 99. Pyrrolysine.
Le codage de fM et celui de SeC ont ceci en commun qu'ils sont l'un et l'autre associés à une extrémité du cadre de lecture : fM au codon initiateur et SeC au codon UAG qui agit le plus souvnt comme codon stop. De plus, fM contient du soufre et SeC est associé à C qui renferme du soufre; SeC contient du sélénium homologue du soufre.
Enfin, l'apparition de fM et celle de SeC dépendent du contexte : éléments en aval pour SeC, coiffe en amont du codon initiateur pour fM.
Symétries du code cb2+.
Formyl-méthionine et symétries du code cb2+. Si l'on considère la formyl-méthionine comme distincte de la méthionine dans le code mitochondriel cb2 des vertébrés, la figure 72 doit être refaite et ses symétries sont changées. Nous comptons comme paire la case M, fM de la strate 2 qui renferme 2 dégénérescences unité. Voici le résultat, appelé cb2+. Les commentaires accompagnant la figure 72 restent valables sauf un changement : il y a 82 cases paires et non 84. Figure 98.
Pyrrolysine. La pyrrolysine pK est un acide aminé naturel chimiquement relié au pyrrol et à l'acide aminé lysine Lys K.
On a observé son apparition chez Methanosarcina barkeri, Archaea voisine des bacilles, dans certaines protéines en réponse au codon UAG habituellement traduit par un stop. Son apparition dépend du contexte, d'opérations "de contrebande": des codons en amont et peut-être en aval du codon UAG dans l'ARNm. Elle apparaîtrait consécutivement àune modification de la lysine fixée sur l'ARNt avant son entrée dans le ribosome. (Gerritsen).
Les bacilles sont réputés suivre le code cb1. Voici donc une 2e addition à ce code devenu cb1+.
Sa place est toute indiquée dans la même case que la lysine K.
Sommaire sur les dérogations de 2e classe. La grille des strates 1, 2, 4 fournit un cadre commode pour présenter les dérogations de 2e classe, comprenant 3 acides aminés naturels supplémentaires en plus d'un hypothétique. Figure 100.
...............
sélénocystéine...............
formyl-méthionine...............
pyrrolysine, (tellurocystéine)Ces dérogations touchent les codes cb1+ et cb2+. Elles intéressent, dans la 4e strate, les 2 quadrants NE et SE qui sont les plus peuplés de dérogations de 1re classe et ajoutent à 2 cases de ces dernières. Elles intéressent les 3 lettres U, A et G à l'exclusion de C.
Co-traductions, acides aminés non-naturels En faisant usage du codon UAG chez Escherichia coli, chez les eucaryotes et les eubactéries, on a pu insérer dans une chaîne protéique de nombreux acides aminés artificiels, leur nombre dépassant la centaine. Le processus est qualifié de co-traductionnel. Référence Schultz et Wang.
Références.
http://www.nature.com/cgi-taf/DynaPage.taf?file=/nbt/journal/v19/n6/full/nbt0601_532.html Wang
http://www.molbiol.umu.se/forskning/persson/
http://www.nature.com/cgi-taf/DynaPage.taf?file=/nbt/journal/v19/n6/full/nbt0601_532.html
insertion de O-méthyl-L-tyrosine Om Tyr non-naturelle, codon UAG
cf Schultz
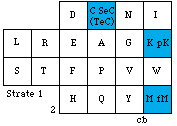
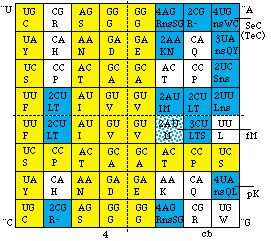
Fig. 100. Dérogations de 2e classe ajoutées à celle de 1re classe. 3 acides aminés naturels supplémentaires et un hypothétique : pK provenant de UAG, fM, de AUG, SeC, de UGA (et TeC, qui pourrait provenir de UGA). Strate 4 : les nombres sont les dégénérescences résultantes des codons.
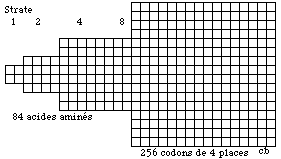
Fig. 101. Strates 1, 2, 4, 8. Logique 22k.
Dérogations de 3e classe.
Alphabet de 4 lettres.
Codons de 3 places.
De l'alphabet de 4 lettres UCAG et de codons de 3 places, on n'a jamais obtenu que les 64 codons connus.Codons de 4 places. On a réussi à expérimenter avec des codons de 4 places dans Escherichia coli : UAGA codant pour un résidu naturel Leucine. Escherichia coli est réputée suivre le code normal des codons à 3 places.
Il y aurait alors en principe 4 cases remplaçant chaque case d'une strate 4 normale de 64 cases, pour donner une strate 8 ayant 256 cases.
4*4*4*4 = 256
Dans une logique 22k il y aurait donc la possibilité théorique de 256 codons et celle de 84 acides aminés dans les cases des strates 1, 2, 4. Figure101.
Référence.
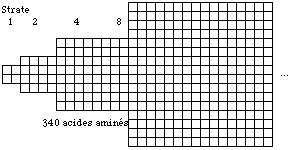
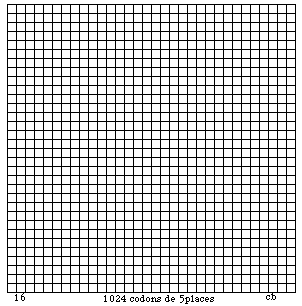
Fig. 102. Strates 1, 2, 4, 8, 16. Logique 22k.
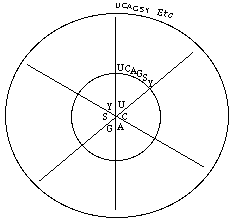
Fig. 103. Logique rayonnante passe-partout pour former des mots de 3 lettres à partir d'un alphabet de 6 lettres. (216 mots).
Codons de 5 places. On a manipulé des codons de 5 places contenant CGGXX, traduits par des acides aminés non naturels. X représente l'une des bases UCAG. Le codon 3 places CGG seul code pour l'arginine R. La case CGG est remplacée par 16 cases.
Il y aurait alors 16 cases remplaçant chaque case d'une strate 4 de 64 cases, pour donner une strate 16 ayant 1024 cases .
4*4*4*4*4 = 1024
Dans une logique 22k. il y aurait donc théoriquement possibilité de 1024 codons et de 340 acides aminés dans les 340 cases des strates 1, 2, 4 et 8. Figure 12.
Les auteurs mentionnent que 16 acides aminés sont possibles et en nomment 2 :
....................
p nitrophényl alanine (nitro Phe)....................
l nitrobenzoxydiazolyl L-lysine [Lysine(NBD)]
Dérogations de 3e classe.
Alphabets de 6 lettres.
Alphabet UCAGxy .
Voici les bases supplémentaires x et y.....................
2-amino-6-(N,N- diméthylamino) purine (x)....................
2-amino-6-(N,N- diméthylamino) pyridine-2-1 (y)Alphabet de 6 lettres UCAGsy. Voici les bases supplémentaires s et y.
....................
2-amino-6-(2-thiény1) purine (s)....................
2-amino-6-(N,N- diméthylamino) pyridine-2-1 (y)Codons de 3 places. Les acides aminés produits sont non naturels. La grille du québécium n'offre pas de cadre commode d'affichage des codons possibles de 3 places établis à partir d'un alphabet de 6 lettres. Selon la combinatoire, pour chacun des alphabets ci-dessus, il y a 216 mots ou codons de 3 places possibles.
6*6*6 = 216
Une grille rayonnante à 6 secteurs passe-partout permet de les afficher. La grille rayonnante à un nombre arbitraire de secteurs est une sorte de généralisation de la grille du québécium 4e strate qui en a 4.
Les bases supplémentaires sont correspondantes l'une de l'autre, étant l'une purique et l'autre pyrimidique. On peut imaginer que cette complémentarité imposerait des restrictions aux codons réalisables.
Il n'y a pas de conjecture évidente à formuler sur le nombre d'acides aminés, sauf qu'il serait plus grand que 20.
Références.
http://www.mindfully.org/GE/GE2/ Invading-Genetic-Code-AAAS.htm
http://arxiv.org/PS_cache/cond-mat/pdf/0204/0204044.pdf hypercube
http://www.lemonde.fr/article/0,5987,3244--264877-,00.html
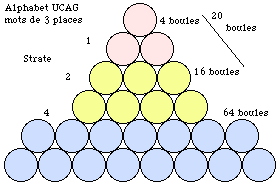
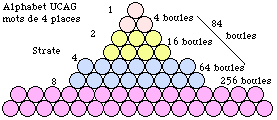
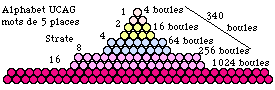
Fig. 104. Le tétraèdre brisé dans la logique 22k pour un alphabet de 4 lettres et des codons de 3, 4 ou 5 places.
Représentations tridimensionnelles.
Les codes de ce chapitre sauf celui issu de l'alphabet de 6 lettres peuvent se représenter dans des tétraèdres brisés ou non ayant l'aspect des figures 64 et 65 qui précèdent, en remplaçant les cases planes par des boules de même contenu. Les figures ne montrent pas le contenu des boules. Elles résument la discussion sur les nombres théoriques dans les dérogations de 3e classe. Figure 104.
4e classe de dérogations.
Les vivants seraient-ils capables de synthétiser des protéines en dehors de la traduction codée par un code biomoléculaire avec ARN, codons, anticodons etc? Afin de n'ignorer aucun type de dérogation, même potentielle, au code biomoléculaire normal, il est opportun de se poser cette question, car il y aurait là une 4e classe de dérogations. Les riboenzymes, dont l'existence a été découverte récemment, contiennent de l'ARN et les enzymes sont capables de synthétiser des acides aminés mais non de les assembler en protéines.
Dans le cas où de telles dérogations existeraient, on peut se demander si le système du québécium serait capable de les représenter.
Mais toute la documentation que l'auteur a pu consulter est unanime à exclure cette hypothèse.
Conclusions.
Nous pouvons conclure ce chapitre en constatant que le système du québécium s'adapte aux dérogations des 1re, 2e et 3e classes touchant les acides aminés naturels sans changements ou avec des changements mineurs. Il s'adapte même aux dérogations de 3e classe associées aux acides aminés non-naturels tant que l'alphabet n'a que 4 lettres msis cesse de s'adapter lorsque l'alphabet a 6 lettres.
Isotopies des 20 acides aminés.
Les dérogations de 2e classe peuvent se décrire et nous suggérons qu'elles s'expliquent par le principe d'une isotopie appliquée à 2 acides aminés de compositions chimiques voisines qui sont placés dans une même case de la strate 2 comme suit.
....................
Méthionine et formyl-Méthionine M et fM.....................
Cystéine et Séléno-Cystéine C et SeC.....................
(en plus de la Telluro-cystéine hypothétique TeC).....................
Lysine et pyrrolysine K et pK.Ainsi est préservée la vingtaine des cases sinon des acides aminés naturels. Il reste assurément à expliquer pourquoi ces isotopies se réalisent sélectivement.
L'isotopie chimique expliquant la 2e classe de dérogations peut se mettre en parallèle avec une explication chimique du changement d'affectation d'un codon lors d'une dérogation de 1re classe. Un tel changement d'affectation est évidemment associé à un changement chimique, non du codon lui-même, mais de certains facteurs dans les étapes de sa traduction : anticodon, ARNt, ARNr etc.
Le code biomoléculaire ne montre pas les étapes intermédiaires entre codon et acide aminé. Lorsque l'étape finale n'est pas l'isotope normal, il faut imaginer un empêchement ou une nécessité qui réclame de préférer, à l'isotope normal, l'isotope non-normal. Lorsque c'est la formyl-méthionine qui apparaît, on sait que la "correction" ne se fait pas attendre : le groupement formyl disparaît (et la méthionine disparaît ensuite). On croirait à une sorte d'erreur qu'il faut vite corriger.
Pourquoi 20 acides aminés?
Afin de satisfaire aux symétries que contient le système du québécium.
Référence Bryan Hayes.
... something special about 20 and 64...
http://www.sigmaxi.org/amsci/issues/Comsci98/compsci9801.html
Représentation
en 6 dimensions.
Voici une intéressante représentation du code génétique normal, cb1, contenu de la strate 4, par un hypercube Booléen en 6 dimensions. Figure 105.
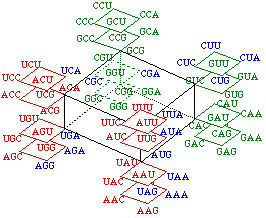
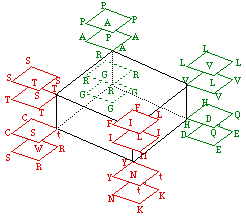
Fig. 105. Voici une intéressante représentation du code génétique normal, cb1, contenu de la strate 4, par un hypercube Booléen en 6 dimensions. D'après Jimenez-Monta~no et al. http://arxiv.org/cond-mat/pdf/0204/0204044.pdf
http://arxiv.org/abs/cond-mat/0204044
Codons et leurs traductions aux 8*8 sommets. Les codons malléables mis en évidence.
Les codons mallébles ne se trouvent pas particulièrement rassemblés dans ce mode de regroupement 8 par 8. Ils sont mieux rassemblés dans le regroupement 16 par 16 du présent système aux figures 92 et 100.
La langue
française. Un alphabet
condensé.
Tableau 40. L'alphabet condensé 20 lettres.
1 A
2 B
C par S et K
3 D
4 E
5 F
6 G
H nil
7 Iy
8 J
9Kcq
10 L
11 M
12 N
13 Ow
14 P
Q parK
15 R
16 Scx
17 T
18 Uw
19 Vw
W par V, O et U
X par K et S
Y par I
20 Z
Il reste 20 lettres dont 5 ont des isotopes. Silence et ponctuation sont en sus. Figure 106.
Le parallèle se présente : 20 lettres dans l'alphabet de la langue française ainsi condensé, et 20 acides aminés dans les strates 1 et 2 du code biomoléculaire. Pourquoi 20 dans les 2 cas? La question est reprise au Chapitre 18.
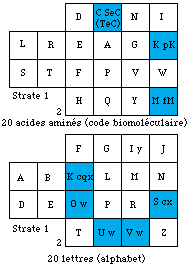
Fig. 106. Alphabet 20 lettres placé dans les strates 1 et 2. Le code cb pour comparaison. Isotopies dans les 2 cas.
Système du Québécium
Fin de
Chapitre 5. Applications aux biomolécules. Les dérogations au code biomoléculaire normal.
QbSyst2e.9.html![]()
Système du Québécium
Suivant QbSyst2e.10.html
Précédent QbSyst2e.8.html
Début QbSyst2e.1.html